Accueil - À propos - Plateformes de recherche - Centre de protéomique clinique
Le Centre de protéomique clinique de l’Hôpital général juif englobe le Centre de protéomique du cancer Segal (SCPC) et le programme Warren Y. Soper de protéomique translationnelle. Le SCPC applique des méthodes de pointe en protéomique et en métabolomique basées sur la spectrométrie de masse quantitative pour la recherche, fournit des services aux groupes universitaires, pharmaceutiques et biotechnologiques, et transpose les essais de spectrométrie de masse quantitative en clinique
Identification des PTM
Protéomique quantitative relative et phosphoprotéomique
Protéomique quantitative ciblée par SM et métabolomique
MultiOMICS
Interactions protéine-
protéineNos technologies peuvent être appliquées à une large gamme d’échantillons, des protéines et complexes protéiques purifiés aux biofluides, cellules, biopsies et tissus FFPE, ne nécessitant que des quantités minimales d’échantillons (par exemple, quelques µL de sang; quelques µg de protéines totales provenant de cellules et de tissus).
Le Centre de protéomique du cancer Segal fait partie de deux réseaux pancanadiens, le TMIC (The Metabolomics Innovation Centre) et le PCPC (Pan-Canadian Proteomics Centre).
Nous développons de nouvelles méthodes et stratégies pour la protéomique quantitative (découverte et ciblée) et la métabolomique (ciblée) en mettant l’accent sur la robustesse, la précision et le débit. Cela comprend la quantification à l’échelle du système des changements relatifs de jusqu’à 10 000 protéines et sites de phosphorylation dans différents échantillons, ainsi que le développement de tests ciblés pour la quantification précise et absolue des concentrations de protéines ou des stœchiométries de phosphorylation. En utilisant des peptides standard marqués aux isotopes stables (SIS), nous sommes en mesure de quantifier de manière absolue les concentrations endogènes de 274 protéines dans le plasma humain à partir de moins de 10 µL de sang.
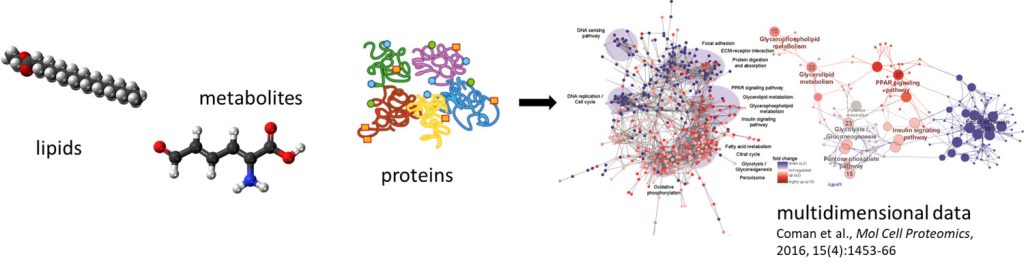
Dans le cadre de l’Institut Lady Davis pour la recherche biomédicale, nous menons des projets de recherche fondamentale pour améliorer la compréhension, le diagnostic et le traitement du cancer afin d’améliorer la médecine de précision. Nous appliquons des approches multiOMICS qui permettent la quantification parallèle du (phospho)protéome, du métabolome et du lipidome à partir de quantités minimales d’échantillons. Nous appliquons la (phospho)protéomique quantitative à la découverte, à l’échelle du système, de changements spécifiques à la maladie et au traitement, et nous développons des stratégies sur mesure pour améliorer la quantification de marqueurs et de voies spécifiques du cancer par spectrométrie de masse.
Dans le cadre de la médecine de précision, les tests génétiques sont devenus un facteur déterminant dans l’orientation des thérapies anticancéreuses, comme le statut de mutation de l’EGFR dans le cancer du poumon non à petites cellules. Cependant, les taux de réponse à ces thérapies guidées sont souvent modestes. Cela peut être dû à la discordance entre le génome et le phénotype réel, qui est reflété par les niveaux d’expression des protéines cancéreuses (mutées) et l’activité des voies de signalisation pertinentes. Par conséquent, notre objectif est de compléter les données génomiques par des informations systémiques sur l’expression des protéines et les états de phosphorylation des protéines à partir de biopsies tissulaires, ce qui permettra d’identifier les causes de la résistance imprévue aux traitements et d’améliorer la sélection des traitements appropriés.
Nous proposons une gamme de services tels que la préparation des échantillons, la mesure par spectrométrie de masse ainsi que l’analyse et la présentation des données. Nos méthodes peuvent être appliquées à une grande variété d’échantillons, y compris des protéines purifiées et des complexes protéiques, des cellules, des tissus et des biofluides.
Nous proposons l’identification par spectrométrie de masse des protéines et des PTM dans un échantillon donné, notamment l’acétylation, la glycosylation, la phosphorylation, le clivage protéolytique et l’ubiquitination. Seuls les résultats de haute confiance sont rapportés en utilisant des outils et des stratégies d’analyse de données de pointe.
L’analyse approfondie des cellules, des tissus et des biofluides conduira à l’identification des protéines et des voies régulées. Selon le type d’échantillon, il est possible de quantifier jusqu’à 10 000 protéines et plus de 10 000 sites de phosphorylation à partir de seulement 25 µg (protéome) ou 100 µg (phosphoprotéome) de protéines totales par lysat. Seules les identifications à haut degré de confiance réalisées à l’aide d’outils et de stratégies d’analyse de données de pointe sont rapportées. Les protéines et les sites de phosphorylation significativement régulés seront signalés à l’aide de seuils de changement de facteur basés sur la distribution des données et les tests statistiques. La visualisation des voies régulées, etc., est disponible sur demande.
Nous avons mis au point un grand nombre de tests pour la quantification absolue précise de protéines sélectionnées en utilisant des peptides standard de référence marqués par des isotopes stables (SIS) en combinaison avec la spectrométrie de masse ciblée (Suivi de réactions multiples, MRM ; Suivi de réactions parallèles, PRM).
Ces essais hautement spécifiques permettent la détermination précise et sensible des concentrations de protéines en mole par volume/cellule/poids, jusqu’à la gamme attomolaire. La MRM et la PRM peuvent être utilisées pour la quantification à haut débit (multiplexée) des protéines sans avoir besoin d’anticorps. En outre, elles sont plus précises et spécifiques que les Western blots. La SM ciblée utilisant des peptides SIS permet de déterminer les stœchiométries de phosphorylation.
Notre service Multi-OMICS consiste en l’analyse combinée du protéome, du métabolome et du lipidome à partir d’un seul échantillon (pas de réplicats, tout provient d’un seul flacon) en utilisant notre stratégie SIMPLEX (Simultaneous Metabolite, Protein, Lipid Extraction). Les ensembles de données complémentaires permettent de visualiser l’interconnexion des différentes classes biomoléculaires, ce qui donne une vision extrêmement détaillée du phénotype de l’échantillon.
Nous employons la réticulation chimique des protéines pour identifier les partenaires d’interaction et les composants des complexes protéiques par spectrométrie de masse. Cette approche permet également de prédire les structures des protéines qui ne sont pas facilement déterminées par la cristallographie aux rayons X ou d’autres approches « traditionnelles ».
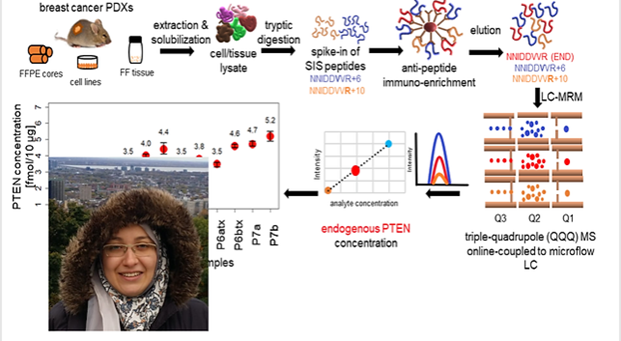
Nos tests immuno-MS sont basés sur l’immuno-enrichissement des cibles à l’aide d’anticorps anti-peptides et anti-protéines, combinés à la spectrométrie de masse pour la quantification hautement sensible des protéines et de leurs PTM, à partir de types d’échantillons cliniquement pertinents (par exemple, des biopsies à l’aiguille). Les tests immuno-MS anti-peptides sont établis pour AKT1, AKT2, et en cours de développement pour P13K, PD-L1 et PTEN (peptide+protéine).

En tant que membre du Metabolomics Innovation Centre (TMIC), nous proposons des tests quantitatifs de métabolomique basés sur la MS pour des centaines de petites molécules, avec une variété de panels représentant différentes voies métaboliques ou classes de molécules.

L’un de nos principaux objectifs est la transposition en clinique de tests ciblés basés sur la SM pour les protéines et les petites molécules. Cela permettra une détection précoce plus sensible et plus précise des maladies, une meilleure optimisation des traitements et un meilleur suivi thérapeutique.
Nous développons des tests spécifiques de spectrométrie de masse à surveillance de réactions multiples (MRM-MS) pour quantifier les petites molécules dans le sang et le plasma, en étroite collaboration avec des cliniciens de l’Hôpital général juif.
Nous développons en outre des tests immuno-MALDI (iMALDI) pour la quantification hautement sensible, spécifique et automatisée des protéines et de leurs états de phosphorylation à partir de cellules, de tissus frais congelés et de tissus FFPE. Le flux de travail comprend l’extraction des protéines, la digestion enzymatique, l’ajout de peptides standard de référence marqués aux isotopes stables (SIS) et l’immunocapture combinée du peptide endogène et de son homologue SIS. Le signal est mesuré par MALDI-MS, ce qui permet la quantification absolue des protéines. Comme le flux de travail ne nécessite aucun système LC et peut être entièrement automatisé, il représente une technologie prometteuse pour quantifier précisément les protéines à partir de quantités minimes d’échantillons, comme les biopsies à l’aiguille, avec la robustesse requise pour un cadre clinique réel.
Comment iMALDI peut améliorer les diagnostics cliniques. Popp, R., Basik, M., Spatz, A., Batist, G., Zahedi, R.P., Borchers, C.H., 2018. Analyseur Clin. Chem. 64, 1271-1272. 143, 2197-2203.


Le Dr Borchers est professeur au département d’oncologie Gerald Bronfman de l’Institut Lady Davis de l’Hôpital général juif de l’Université McGill, et titulaire de la chaire Segal McGill en oncologie moléculaire. Il est devenu membre de l’Académie canadienne des sciences de la santé en 2013 et a reçu le Life Sciences BC Award/Genome BC Award for Scientific Excellence en 2016. Ses recherches englobent la protéomique structurelle et quantitative ainsi que la métabolomique quantitative, avec un accent clair sur les applications et la traduction cliniques. Le laboratoire du Dr Borchers a mis au point des tests de spectrométrie de masse ciblés pour la quantification » absolue » de milliers de protéines provenant de cellules, de tissus et de biofluides, notamment des membres de la voie PI3K/AKT/mTOR et de l’axe de signalisation PD-L1. Il a également mis au point des tests de SM pour déterminer les taux de mutation de facteurs cancéreux connus dans les tissus tumoraux au niveau des protéines, et pour déterminer la concentration d’immunothérapies dans le sang des patients, pour le suivi des médicaments. Les tests MS ciblés développés dans le laboratoire de Borchers sont utilisés à la fois pour la recherche fondamentale et pour compléter les tests génomiques existants en oncologie de précision. Le Dr Borchers a publié plus de 325 articles sur la protéomique basée sur la spectrométrie de masse, avec un indice H de 77 et plus de 25 000 citations.

(Protéomique structurelle, MS clinique, Métabolomique). Il a obtenu son doctorat en médecine au Second Institut médical de Moscou, Moscou, Russie, et son doctorat en chimie bioorganique à l’Institut de chimie bioorganique de l’Académie nationale des sciences du Belarus, Minsk, Belarus. Il a effectué des études postdoctorales au National Institute of Environmental Health Sciences, National Institutes of Health à Research Triangle Park, NC et à l’Université de Caroline du Nord à Chapel Hill, NC. Il a été professeur assistant de recherche à l’Université de Victoria-Genome BC Proteomics Centre, Université de Victoria, Colombie-Britannique, Canada. Ses recherches portent sur le développement et l’application des approches de la protéomique structurelle pour étudier la structure des protéines dans des pathologies telles que les maladies neurodégénératives de mauvais repliement des protéines. Depuis peu, il est impliqué dans le développement et l’application des méthodes analytiques MS en clinique et dans le développement d’analyses métabolomiques.

Yassene est professeur adjoint au Centre de protéomique et de métabolomique du Centre médical de l’université de Leiden, aux Pays-Bas. Il est également responsable de la bioinformatique au Proteomics Centre de l’université de Victoria, au Canada, où il est également professeur adjoint à la division des sciences médicales. Il a obtenu son doctorat à l’université de Göttingen, en Allemagne. Une grande partie des activités récentes de Yassene se concentre sur la protéomique quantitative ciblée et ses applications cliniques. Il a coécrit de nombreux ouvrages sur les méthodes et les applications de la quantification précise des protéines du plasma sanguin à l’aide de la spectrométrie de masse et de normes internes de peptides de substitution lourdement marqués. Il est également actif dans la recherche de nouvelles approches pour l’analyse des données de spectrométrie de masse, l’intégration de la -omique, ainsi que l’automatisation de l’extraction d’informations de ces données.

Adriano est coordinateur administratif principal au SCPC. Il a été professeur associé à l’Université fédérale de Minas Gerais (UFMG), au Brésil, pendant 18 ans avant de s’installer au Canada en 2022 et de rejoindre le SCPC le 22 novembre. Diplômé en sciences biologiques (licence en zoologie) et titulaire d’un doctorat en sciences biologiques (physiologie et pharmacologie) de l’UFMG, Adriano a également été chercheur postdoctoral à l’Université de la Méditerranée (Marseille, France; 1999/2001) et professeur invité à l’Université de l’Alberta (2013-2014). Il possède une solide expérience dans la conduite de projets de recherche sur la découverte de médicaments axés sur la structure ou la fonction, combinant la biochimie, la pharmacologie moléculaire, la biologie cellulaire et les approches protéomiques. Adriano est responsable de la supervision et de la gestion des opérations, des budgets et des activités de la recherche en protéomique et de son équipe au SCPC.

Le Dr Richard est chercheur associé au SCPC et chef de projet d’un projet GAPP financé par Génome Canada. Il a rejoint le SCPC en 2015. Il développe des méthodes de protéomique quantitative (découverte et validation) ainsi que de métabolomique (quantification ciblée de métabolites). Il est impliqué dans de multiples projets collaboratifs au sein de l’Institut Lady Davis et de l’Université McGill. Vincent a terminé son doctorat en 2014 dans le laboratoire du Dr Vladimir Titorenko où il a utilisé la protéomique, la lipidomique et la métabolomique basées sur la LC/MS pour élucider les mécanismes moléculaires régulant le vieillissement et la durée de vie d’organismes modèles.

Deema is an experienced scientist working at the SCPC. She obtained her bachelor’s degree in Pharmacy from the Jordan University of Science and Technology. She later received a Master of Medical Science in Clinical Biochemistry from the University of Calgary. Before joining the SCPC, Deema held positions as a Laboratory Scientist at Jordan University of Science and Technology and Alberta Precision Laboratories, where her research focused on the development of mass spectrometry-based tools for early and accurate diagnosis of metabolic disorders such as Lysosomal Storage Disorders and Congenital Adrenal Hyperplasia using dried blood spots. Since joining the SCPC in 2021, Deema has contributed to the institute’s research, method development and validation and collaborative projects in clinical mass spectrometry, metabolomics, lipidomics, and proteomics.

Dennis is a research technician at the SCPC. Dennis graduated with a bachelor’s degree in Biological Sciences at the University of British Columbia. He worked as a laboratory assistant at the University of British Columbia under Dr. Shyh-Dar Li in the pharmaceutical sciences department, assisting with nanoparticle research and drug delivery projects. Dennis joined the SCPC in 2022, to aid in analytical mass spectrometry, metabolomics, and proteomics research.

Ute a rejoint le Centre de protéomique du cancer Segal en 2019. Elle a obtenu sa licence en chimie à l’Académie des sciences naturelles et techniques d’Isny, en Allemagne.


Georgia Mitsa is a PhD candidate in the Department of Experimental Medicine at McGill University in Montreal, Canada. Her research combines proteomics, metabolomics and genomics to gain mechanistic insights into hard-to-treat malignancies. Georgia aims to advance the translation of multi-omics into the clinic to generate a complete phenotypic picture of oncogenic molecular changes. The ultimate goal is to enable quick and reliable clinical decision-making using multi-omics analysis of clinical biopsies. To this end, Georgia has developed several quantitative mass spectrometry-based methods for multi-omics molecular characterization of biobanked clinical samples. She is currently spearheading a large-scale study to investigate the molecular changes in metastatic colorectal cancers that are refractory to standard-of-care treatment. She further initiated and conducts a large-scale study targeting proteomic changes in pre-invasive breast ductal carcinoma; this project applies her recently-published method for mass spectrometry-based quantitative proteomics from core-needle biopsies embedded in formalin and paraffin.

Connie is a PhD candidate who joined the SCPC in 2017. She is supervised by Dr. Gerald Batist. Her project involves validating and applying quantitative proteomics technologies in cancer tissue samples to better predict patients’ individual responses to targeted treatments.

Vincent est un étudiant diplômé qui a rejoint le SCPC en 2019. Il est cosupervisé par le Dr Alan Spatz, et il travaille sur un test immuno-MS pour quantifier l’axe PD-1/PD-L1 afin de mieux prédire la réponse des tumeurs individuelles aux inhibiteurs de points de contrôle immunitaire. Il a complété son baccalauréat en pharmacologie (programme coopératif) et sa maîtrise à l’Université de Sherbrooke avant de se joindre au SCPC. Tout au long de ses études, il s’est attaché à comprendre la variabilité de l’efficacité des traitements, en particulier pour le cancer. Cela l’a amené à explorer la protéomique basée sur la spectrométrie de masse pour mieux comprendre la biologie des tumeurs.

Neda est une étudiante diplômée qui a rejoint le SCPC en 2022. Elle est co-supervisée par le Dr Allen Spatz, et elle travaille sur des essais immuno-Ms pour étudier la signalisation PD-L1.

Forough est une étudiante diplômée qui a rejoint le SCPC en 2022. Elle a obtenu un diplôme de médecine à l’Université Beheshti des sciences médicales (SBMU) et travaille sur la protéomique structurelle.

Costa est un étudiant diplômé qui a rejoint le SCPC en 2022 et il travaille sur les approches bioinformatiques dans la recherche sur le cancer.

Fatemeh is a graduate student who has joined the SCPC in 2022. She is co-supervised by Dr. Michael Pollak, and she is working on Characterization of Signaling Pathway Activation in Breast Cancer using Comprehensive Targeted Phosphoproteomics.

Negarsadat Mostolizadeh est étudiante en maîtrise à l’Université McGill et est devenue membre du SCPC en 2022. Elle est co-supervisée par le Dr. Mark Basik. Son projet de recherche actuel porte sur l’utilisation de l’essai MRM pour quantifier l’expression de Her2 dans les tumeurs à faible teneur en Her2. Le but de ce travail est de distinguer les cas HER2-low des cas HER2-négatifs et de fournir de meilleures informations pour la prise de décision thérapeutique pour les patients atteints de cancer.

Neginsadat Mostolizadeh est étudiante en maîtrise à l’Université McGill et membre du SCPC. Elle a rejoint l’équipe en 2022 et est co-supervisée par le Dr Gerald Batist. Son projet de thèse de maîtrise porte sur le test et l’optimisation de la performance d’un profil protéomique associé à la réponse de Capivasertib dans les lignées cellulaires cancéreuses et les échantillons de tumeurs de patients. Son objectif ultime est de rendre les données du profil protéomique utiles dans les milieux cliniques.
Qasrawi DO, Al-Ghabkari A, Khan RM, Petrotchenko EV, Montero-Odasso M, Borchers CH.
A Simplified Proteomics LC-MRM-MS Assay for Determination of apoE Genotypes in Plasma Samples.
J Proteome Res. 2024 Feb 27. DOI: 10.1021/acs.jproteome.3c00557. Online ahead of print.
PMID: 38412507.
Luo V, Shen C, Worme S, Bhagrath A, Simo-Cheyou E, Findlay S, Hebert S, Poon W, Aryanpour Z, Zhang T, Zahedi R, Boulais J, Buchwald Z, Borchers CH, Cote J, Kleinman C, Mandl J, Orthwein A.
The Deubiquitylase Otub1 Regulates the Chemotactic Response of Splenic B Cells by Modulating the Stability of the γ-Subunit Gng2.
Molecular and Cellular Biology 2024 Jan 29; 44:1, 1-16, DOI: 10.1080/10985549.2023.2290434.
Mohammed Y, Tran K, Carlsten C, Ryerson C, Wong A, Lee T, Cheng MP, Vinh DC, Lee TC, Winston BW, Sweet D, Boyd JH, Walley KR, Haljan G, McGeer A, Lamontagne F, Fowler R, Maslove D, Singer J, Patrick DM, Marshall JC, Murthy S, Jain F, Borchers CH, Goodlett DR, Levin A, Russell JA; ARBs CORONA I Consortium.
Proteomic Evolution from Acute to Post-COVID-19 Conditions.
J Proteome Res. 2024 Jan 5;23(1):52-70. DOI: 10.1021/acs.jproteome.3c00324.
PMID: 38048423 Free PMC article.
Sobsey CA, Mady N, Richard VR, LeBlanc A, Zakharov T, Borchers CH, Jagoe RT.
Measurement of CYP1A2 and CYP3A4 activity by a simplified Geneva cocktail approach in a cohort of free-living individuals: a pilot study.
Front Pharmacol. 2024 Feb 2;15:1232595. DOI: 10.3389/fphar.2024.1232595. eCollection 2024.
PMID: 38370474 Free PMC article.
Michaud SA, Pětrošová H, Sinclair NJ, Kinnear AL, Jackson AM, McGuire JC, Hardie DB, Bhowmick P, Ganguly M, Flenniken AM, Nutter LMJ, McKerlie C, Smith D, Mohammed Y, Schibli D, Sickmann A, Borchers CH.
Multiple reaction monitoring assays for large-scale quantitation of proteins from 20 mouse organs and tissues.
Commun Biol. 2024 Jan 2;7(1):6. DOI: 10.1038/s42003-023-05687-0.
PMID: 38168632.
Reboucas P, Fillebeen C, Botta A, Cleverdon R, Steele AP, Richard VR, Zahede RP, Borchers CH, Burelle Y, Hawkes TJ, Pantopoulos K, Sweeney G.
Discovery-Based Proteomics Identify Skeletal Muscle Mitochondrial Alterations as an Early Metabolic Defect in a Mouse Model of β-Thalassemia
Int. J. Mol. Sci. 2023 Feb 23, 24(5), 4402; https://doi.org/10.3390/ijms24054402.
Mohammed Y, Goodlett D, Borchers CH.
Bioinformatics Tools and Knowledgebases to Assist Generating Targeted Assays for Plasma Proteomics
Methods Mol Biol. 2023 Feb 12; 2628: 557-577. doi: 10.1007/978-1-0716-2978-9_32.
PMID: 36781806
Mohammed Y, Goodlett D, Borchers CH.
Absolute Quantitative Targeted Proteomics Assays for Plasma Proteins
Methods Mol Biol. 2023 Feb 12; 2628: 439-473. doi: 10.1007/978-1-0716-2978-9_27.
PMID: 36781801
Prabhu SA, Moussa O, Gonçalves C, LaPierre JH, Chou H, Huang F, Richard VR, Ferruzo PYM, Guettler EM, Soria-Bretones I, Kirby L, Gagnon N, Su J, Silvester J, Krisna SS, Rose AAN, Sheppard KE, Cescon DW, Mallette FA, Zahedi RP, Borchers CH, Del Rincon SV, Miller WH.
Inhibition of the MNK1/2-eIF4E Axis Augments Palbociclib-Mediated Antitumor Activity in Melanoma and Breast Cancer
Mol Cancer Ther, 2023 Feb 1;22(2):192-204. doi: 10.1158/1535-7163.MCT-22-0092.
PMID: 36722142
Preston SEJ, Richard VR, Del Rincón SV, Borchers CH, Zahedi RP.
Proteomic Assessment of the Murine Mammary Gland Extracellular Matrix.
Methods Mol Biol, 2023;2614:261-271. doi: 10.1007/978-1-0716-2914-7_16.
PMID: 36587130
Parra ALC, Freitas CDT, Souza PFN, von Aderkas P, Borchers CH, Beattie GA, Silva FDA, Thornburg RW.
Ornamental tobacco floral nectar is a rich source of antimicrobial peptides.
Plant Sci, 2022 Nov;324:111427. doi: 10.1016/j.plantsci.2022.111427. Epub 2022 Aug 23.
PMID: 36007629
Richard VR, Gaither C, Popp R, Chaplygina D, Brzhozovskiy A, Kononikhin A, Mohammed Y, Zahedi RP, Nikolaev EN, Borchers CH.
Early Prediction of COVID-19 Patient Survival by Targeted Plasma Multi-Omics and Machine Learning.
Mol Cell Proteomics, 2022 Oct;21(10):100277. doi: 10.1016/j.mcpro.2022.100277. Epub 2022 Aug 3.
PMID: 35931319
Smith-Byrne K, Cerani A, Guida F, Zhou S, Agudo A, Aleksandrova K, Barricarte A, Rodríguez-Barranco M, Borchers CH, Gram IT, Han J, Amos CI, Hung RJ, Grankvist K, Nøst TH, Imaz L, Chirlaque-López MD, Johansson M, Kaaks R, Kühn T, Martin RM, McKay JD, Pala V, Robbins HA, Sandanger TM, Schibli D, Schulze MB, Travis RC, Vineis P, Weiderpass E, Brennan P, Johansson M, Richards JB.
Circulating Isovalerylcarnitine and Lung Cancer Risk: Evidence from Mendelian Randomization and Pre-Diagnostic Blood Measurements.
Cancer Epidemiol Biomarkers Prev, 2022 Jul 15:EPI-21-1033. doi: 10.1158/1055-9965.EPI-21-1033. Online ahead of print.
PMID: 35839461
Preston SEJ, Bartish M, Richard VR, Aghigh A, Gonçalves C, Smith-Voudouris J, Huang F, Thebault P, Cleret-Buhot A, Lapointe R, Légaré F, Postovit LM, Zahedi RP, Borchers CH, Miller WH Jr, Del Rincón SV.
Phosphorylation of eIF4E in the stroma drives the production and spatial organisation of collagen type I in the mammary gland.
Matrix Biol, 2022 Jul 13:S0945-053X(22)00092-0. doi: 10.1016/j.matbio.2022.07.003. Online ahead of print.
PMID: 35842012
Pastushkova LK, Goncharov IN, Koloteva MI, Goncharova AG, Kashirina DN, Nosovsky AM, Glebova TM, Kononikhin AS, Borchers CH, Nikolaev EN, Larina IM.
Characteristics of blood plasma proteome changes associated with the hemorrhagic purpura of cosmonauts on the first day after long-term space missions.
Life Sci Space Res (Amst), 2022 May;33:7-12. doi: 10.1016/j.lssr.2022.01.001. Epub 2022 Jan 14.
PMID: 35491032
Mitsa G, Guo Q, Goncalves C, Preston SEJ, Lacasse V, Aguilar-Mahecha A, Benlimame N, Basik M, Spatz A, Batist G, Miller WH Jr, Del Rincon SV, Zahedi RP, Borchers CH.
A Non-Hazardous Deparaffinization Protocol Enables Quantitative Proteomics of Core Needle Biopsy-Sized Formalin-Fixed and Paraffin-Embedded (FFPE) Tissue Specimens.
Int J Mol Sci, 2022 Apr 18;23(8):4443. doi: 10.3390/ijms23084443.
PMID: 35457260
Laselva O, Petrotchenko EV, Hamilton CM, Qureshi Z, Borchers CH, Young RN, Bear CE.
A protocol for identifying the binding sites of small molecules on the cystic fibrosis transmembrane conductance regulator (CFTR) protein.
STAR Protoc, 2022 Apr 7;3(2):101258. doi: 10.1016/j.xpro.2022.101258. eCollection 2022 Jun 17.
PMID: 35434660
Mohammed Y, Touw CE, Nemeth B, van Adrichem RA, Borchers CH, Rosendaal FR, van Vlijmen BJ, Cannegieter SC.
Targeted proteomics for evaluating risk of venous thrombosis following traumatic lower-leg injury or knee arthroscopy.
J Thromb Haemost, 2022 Mar;20(3):684-699. doi: 10.1111/jth.15623. Epub 2022 Jan 6.
PMID: 34919779
Gaither C, Popp R, Zahedi RP, Borchers CH.
Multiple reaction monitoring-mass spectrometry enables robust quantitation of plasma proteins regardless of whole blood processing delays that may occur in the clinic.
Mol Cell Proteomics, 2022 May;21(5):100212. doi: 10.1016/j.mcpro.2022.100212. Epub 2022 Feb 17.
PMID: 35182769
Jennings MJ, Kagiava A, Vendredy L, Spaulding EL, Stavrou M, Hathazi D, Grüneboom A, De Winter V, Gess B, Schara U, Pogoryelova O, Lochmüller H, Borchers CH, Roos A, Burgess RW, Timmerman V, Kleopa KA, Horvath R.
NCAM1 and GDF15 are biomarkers of Charcot-Marie-Tooth disease in patients and mice.
Brain, 2022 Nov 21;145(11):3999-4015. doi: 10.1093/brain/awac055.
PMID: 35148379
Brzhozovskiy A, Kononikhin A, Bugrova AE, Kovalev GI, Schmit PO, Kruppa G, Nikolaev EN, Borchers CH.
The Parallel Reaction Monitoring-Parallel Accumulation-Serial Fragmentation (prm-PASEF) Approach for Multiplexed Absolute Quantitation of Proteins in Human Plasma.
Anal Chem, 2022 Jan 18. doi: 10.1021/acs.analchem.1c03782. Online ahead of print.
PMID: 35040635
Petrotchenko EV, Borchers CH.
Protein Chemistry Combined with Mass Spectrometry for Protein Structure Determination.
Chem Rev, 2022 Apr 27;122(8):7488-7499. doi: 10.1021/acs.chemrev.1c00302. Epub 2021 Dec 30.
PMID: 34968047
Gaspar VP, Ibrahim S, Zahedi RP, Borchers CH.
Utility, Promise, and Limitations of LC-MS based Therapeutic Drug Monitoring in Precision Medicine.
Journal of Mass Spectrometry, 2021 Nov;56(11):e4788. doi: 10.1002/jms.4788. Epub 2021 Nov 4.
PMID: 34738286 Review
Froehlich BC, Popp R, Sobsey CA, Ibrahim S, LeBlanc A, Mohammed Y, Buchanan M, Aguilar-Mahecha A, Pötz O, Chen MX, Spatz A, Basik M, Batist G, Zahedi RP, Borchers CH.
A multiplexed, automated immuno-matrix assisted laser desorption/ionization mass spectrometry assay for simultaneous and precise quantitation of PTEN and p110α in cell lines and tumor tissues.
Analyst, 2021 Oct 25, 146, 6566-6575 doi: 10.1039/d1an00165e. Epub 2021 Sep 29.
PMID: 34585690
Serpa JJ, Popov KI, Petrotchenko EV, Dokholyan NV, Borchers CH.
Structure of prion β-oligomers as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations.
Proteomics, 2021 Sep 5:e2000298. doi: 10.1002/pmic.202000298. Online ahead of print.
PMID: 34482645
Ibrahim S, Lan C, Chabot C, Mitsa G, Buchanan M, Aguilar-Mahecha A, Elchebly M, Poetz O, Spatz A, Basik M, Batist G, Zahedi RP, Borchers CH.
Precise Quantitation of PTEN by Immuno-MRM: A Tool to Resolve the Breast Cancer Biomarker Controversy.
Anal Chem, 2021 Aug 10;93(31):10816-10824. doi: 10.1021/acs.analchem.1c00975. Epub 2021 Jul 29.
PMID: 34324311
Gaither C, Popp R, Borchers SP, Skarphedinsson K, Eiriksson FF, Thorsteinsdóttir M, Mohammed Y, Borchers CH.
Performance Assessment of a 125 Human Plasma Peptide Mixture Stored at Room Temperature for Multiple Reaction Monitoring-Mass Spectrometry.
J Proteome Res, 2021 Sep 3;20(9):4292-4302. doi: 10.1021/acs.jproteome.1c00249. Epub 2021 Jul 16.
PMID: 34270269
Skinnider MA, Rogalski J, Tigchelaar S, Manouchehri N, Prudova A, Jackson AM, Nielsen K, Jeong J, Chaudhary S, Shortt K, Gallagher-Kurtzke Y, So K, Fong A, Gupta R, Okon EB, Rizzuto MA, Dong K, Streijger F, Belanger L, Ritchie L, Tsang A, Christie S, Mac-Thiong JM, Bailey C, Ailon T, Charest-Morin R, Dea N, Wilson JR, Dhall S, Paquette S, Street J, Fisher CG, Dvorak MF, Shannon C, Borchers C, Balshaw R, Foster LJ, Kwon BK.
Proteomics portraits reveal evolutionarily conserved and divergent responses to spinal cord injury.
Mol Cell Proteomics, 2021 Jun 12:100096. doi: 10.1016/j.mcpro.2021.100096. Online ahead of print.
PMID: 34129941
Tran JN, Günther OP, Sherwood KR, Fenninger F, Allan LL, Lan J, Sapir-Pichhadze R, Duquesnoy R, Claas F, Marsh SGE, McMaster WR, Keown PA; Genome Canada Transplant Consortium.
High-throughput sequencing defines donor and recipient HLA B-cell epitope frequencies for prospective matching in transplantation.
Commun Biol, 2021 May 14;4(1):583. doi: 10.1038/s42003-021-01989-3.
PMID: 33990681
Laselva O, Qureshi Z, Zeng ZW, Petrotchenko EV, Ramjeesingh M, Hamilton CM, Huan LJ, Borchers CH, Pomès R, Young R, Bear CE.
Identification of binding sites for ivacaftor on the cystic fibrosis transmembrane conductance regulator.
iScience, 2021 May 15;24(6):102542. doi: 10.1016/j.isci.2021.102542. eCollection 2021 Jun 25.
PMID: 34142049
Mohammed Y, Michaud SA, Pětrošová H, Yang J, Ganguly M, Schibli D, Flenniken AM, Nutter LMJ, Adissu HA, Lloyd KCK, McKerlie C, Borchers CH.
Proteotyping of knockout mouse strains reveals sex- and strain-specific signatures in blood plasma.
NPJ Syst Biol Appl, 2021 May 28;7(1):25. doi: 10.1038/s41540-021-00184-8.
PMID: 34050187
Li FKK, Gale RT, Petrotchenko EV, Borchers CH, Brown ED, Strynadka NCJ.
Crystallographic analysis of TarI and TarJ, a cytidylyltransferase and reductase pair for CDP-ribitol synthesis in Staphylococcus aureus wall teichoic acid biogenesis.
J Struct Biol, 2021 Jun;213(2):107733. doi: 10.1016/j.jsb.2021.107733. Epub 2021 Apr 2.
PMID: 33819634
Bhowmick P, Roome S, Borchers CH, Goodlett DR, Mohammed Y.
An Update on MRMAssayDB: A Comprehensive Resource for Targeted Proteomics Assays in the Community.
Proteome Res, 2021 Apr 2;20(4):2105-2115. doi: 10.1021/acs.jproteome.0c00961. Epub 2021 Mar 8.
PMID: 33683131
Mohammed Y, Bhowmick P, Michaud SA, Sickmann A, Borchers CH.
Mouse Quantitative Proteomics Knowledgebase: reference protein concentration ranges in 20 mouse tissues using 5000 quantitative proteomics assays.
Bioinformatics, 2021 Jan 23:btab018. doi: 10.1093/bioinformatics/btab018. Online ahead of print.
PMID: 33483739
Shahid I, Han J, Hardie D, Baig DN, Malik KA, Borchers CH, Mehnaz S.
Profiling of antimicrobial metabolites of plant growth promoting Pseudomonas spp. isolated from different plant hosts.
3 Biotech, 2021 Feb;11(2):48. doi: 10.1007/s13205-020-02585-8. Epub 2021 Jan 11.
PMID: 33489669
Michaud SA, Pětrošová H, Jackson AM, McGuire JC, Sinclair NJ, Ganguly M, Flenniken AM, Nutter LMJ, McKerlie C, Schibli D, Smith D, Borchers CH.
Process and Workflow for Preparation of Disparate Mouse Tissues for Proteomic Analysis.
J Proteome Res, 2021 Jan 1;20(1):305-316. doi: 10.1021/acs.jproteome.0c00399. Epub 2020 Nov 5.
PMID: 33151080
Gaspar VP, Ibrahim S, Sobsey CA, Richard VR, Spatz A, Zahedi RP, Borchers CH.
Direct and Precise Measurement of Bevacizumab Levels in Human Plasma Based on Controlled Methionine Oxidation and Multiple Reaction Monitoring.
ACS Pharmacol Transl Sci, 2020 Nov 13;3(6):1304-1309. doi: 10.1021/acsptsci.0c00134. eCollection 2020 Dec 11.
PMID: 33344903
De Giorgi M, Jarrett KE, Burton JC, Doerfler AM, Hurley A, Li A, Hsu RH, Furgurson M, Patel KR, Han J, Borchers CH, Lagor WR.
Depletion of essential isoprenoids and ER stress induction following acute liver-specific deletion of HMG-CoA reductase.
J Lipid Res, 2020 Dec;61(12):1675-1686. doi: 10.1194/jlr.RA120001006. Epub 2020 Oct 27.
PMID: 33109681
Leitner A, Bonvin AMJJ, Borchers CH, Chalkley RJ, Chamot-Rooke J, Combe CW, Cox J, Dong MQ, Fischer L, Götze M, Gozzo FC, Heck AJR, Hoopmann MR, Huang L, Ishihama Y, Jones AR, Kalisman N, Kohlbacher O, Mechtler K, Moritz RL, Netz E, Novak P, Petrotchenko E, Sali A, Scheltema RA, Schmidt C, Schriemer D, Sinz A, Sobott F, Stengel F, Thalassinos K, Urlaub H, Viner R, Vizcaíno JA, Wilkins MR, Rappsilber J.
Toward Increased Reliability, Transparency, and Accessibility in Cross-linking Mass Spectrometry.
Structure, 2020 Nov 3;28(11):1259-1268. doi: 10.1016/j.str.2020.09.011. Epub 2020 Oct 15.
PMID: 33065067
Richard VR, Zahedi RP, Eintracht S, Borchers CH.
An LC-MRM assay for the quantification of metanephrines from dried blood spots for the diagnosis of pheochromocytomas and paragangliomas.
Anal Chim Acta, 2020 Sep 1;1128:140-148. doi: 10.1016/j.aca.2020.06.020. Epub 2020 Jul 9.
PMID: 32825898
Ibrahim S, Froehlich B, Aguilar-Mahecha A, Aloyz R, Poetz O, Basik M, Batist G, Zahedi RP, Borchers CH.
Using two peptide isotopologues as internal standards for the streamlined quantification of low-abundance proteins by immuno-MRM and immuno-MALDI.
Anal Chem, 2020 Aug 12. doi: 10.1021/acs.analchem.0c02157. Online ahead of print.
PMID: 32786432
Nikolaev EN, Indeykina MI, Brzhozovskiy AG, Bugrova AE, Kononikhin AS, Starodubtseva NL, Petrotchenko EV, Kovalev GI, Borchers CH, Sukhikh GT.
Mass Spectrometric detection of SARS-CoV-2 virus in scrapings of the epithelium of the nasopharynx of infected patients via Nucleocapsid N protein.
J Proteome Res, 2020 Aug 19. doi: 10.1021/acs.jproteome.0c00412. Online ahead of print.
PMID: 32786682
Froehlich BC, Popp R, Sobsey CA, Ibrahim S, LeBlanc AM, Mohammed Y, Aguilar-Mahecha A, Poetz O, Chen MX, Spatz A, Basik M, Batist G, Zahedi RP, Borchers CH.
Systematic Optimization of the iMALDI Workflow for the Robust and Straightforward Quantification of Signaling Proteins in Cancer Cells.
Proteomics Clin Appl, 2020 Jul 8:e2000034. doi: 10.1002/prca.202000034. Online ahead of print.
PMID: 32643306
Kashirina DN, Brzhozovskiy AG, Pastushkova LKh, Kononikhin AS, Borchers CH, Nikolaev EN, Larina IM.
Semiquantitative Proteomic Research of Protein Plasma Profile of Volunteers in 21-Day Head-Down Bed Rest.
Front Physiol, 2020 Jul 24;11:678. doi: 10.3389/fphys.2020.00678. eCollection 2020.
PMID: 32848806
Liu T, Wang RX, Han J, Qiu YL, Borchers CH, Ling V, Wang JS.
Changes in plasma bile acid profiles after partial internal biliary diversion in PFIC2 patients.
Ann Transl Med, 2020 Mar;8(5):185. doi: 10.21037/atm.2020.01.103.
PMID: 32309332
Sobsey CA, Ibrahim S, Richard VR, Gaspar V, Mitsa G, Lacasse V, Zahedi RP, Batist G, Borchers CH.
Targeted and Untargeted Proteomics Approaches in Biomarker Development.
Proteomics. 2020 May;20(9):e1900029. doi: 10.1002/pmic.201900029. Epub 2020 Jan 30.
PMID: 31729135
Review
Gaither C, Popp R, Mohammed Y, Borchers CH.
Determination of the concentration range for 267 proteins from 21 lots of commercial human plasma using highly multiplexed multiple reaction monitoring mass spectrometry.
Analyst, 2020 May 18;145(10):3634-3644. doi: 10.1039/c9an01893j.
PMID: 32255452
Tilburg J, Michaud SA, Maracle CX, Versteeg HH, Borchers CH, van Vlijmen BJM, Mohammed Y.
Plasma Protein Signatures of a Murine Venous Thrombosis Model and Slc44a2 Knockout Mice Using Quantitative-Targeted Proteomics.
Thromb Haemost, 2020 Mar;120(3):423-436. doi: 10.1055/s-0040-1702229. Epub 2020 Mar 5.
PMID: 32135565
Makepeace KAT, Mohammed Y, Rudashevskaya EL, Petrotchenko EV, Voegtle FN, Meisinger C, Sickmann A, Borchers CH.
Improving identification of in-organello protein-protein interactions using an affinity-enrichable, isotopically-coded, and mass spectrometry-cleavable chemical crosslinker.
Mol Cell Proteomics, 2020 Apr;19(4):624-639. doi: 10.1074/mcp.RA119.001839. Epub 2020 Feb 12.
PMID: 32051233
Bhardwaj M, Weigl K, Tikk K, Holland-Letz T, Schrotz-King P, Borchers CH, Brenner H.
Multiplex quantitation of 270 plasma protein markers to identify a signature for early detection of colorectal cancer.
Eur J Cancer, 2020 Mar;127:30-40. doi: 10.1016/j.ejca.2019.11.021. Epub 2020 Jan 21.
PMID: 31972396
Shin JJH, Liu P, Chan LJ, Ullah A, Pan J, Borchers CH, Burke JE, Stefan C, Smits GJ, Loewen CJR.
pH Biosensing by PI4P Regulates Cargo Sorting at the TGN.
Dev Cell, 2020 Feb 24;52(4):461-476.e4. doi: 10.1016/j.devcel.2019.12.010. Epub 2020 Jan 9.
PMID: 31928972
Eshghi A, Pistawka AJ, Liu J, Chen M, Sinclair NJT, Hardie DB, Elliott M, Chen L, Newman R, Mohammed Y, Borchers CH.
Concentration determination of >200 proteins in dried blood spots for biomarker discovery and validation.
Mol Cell Proteomics, 2020 Mar;19(3):540-553. doi:10.1074/mcp.TIR119.001820. Epub 2020 Jan 2.
PMID: 31896676
Silva F, Guirgis A, von Aderkas P, Borchers CH, Thornburg R.
LC-MS/MS based comparative proteomics of floral nectars reveal different mechanisms involved in floral defense of Nicotiana spp., Petunia hybrida and Datura stramonium.
J Proteomics, 2020 Feb 20;213:103618. doi: 10.1016/j.jprot.2019.103618. Epub 2019 Dec 14.
PMID: 31846763
Mohammed Y, Kootte RS, Kopatz WF, Borchers CH, Büller HR, Versteeg HH, Nieuwdorp M, van Mens TE.
The intestinal microbiome potentially affects thrombin generation in human subjects.
J Thromb Haemost, 2020 Mar;18(3):642-650. doi: 10.1111/jth.14699. Epub 2019 Dec 27.
PMID: 31808596
Sobsey CA, Ibrahim S, Richard VR, Gaspar V, Mitsa G, Lacasse V, Zahedi RP, Batist G, Borchers CH.
Targeted and Untargeted Proteomics Approaches in Biomarker Development.
Proteomics, 2020 May;20(9):e1900029. doi: 10.1002/pmic.201900029. Epub 2020 Jan 30.
PMID: 31729135
Makepeace KAT, Brodie NI, Popov KI, Gudavicius G, Nelson CJ, Petrotchenko EV, Dokholyan NV, Borchers CH.
Ligand-induced disorder-to-order transitions characterized by structural proteomics and molecular dynamics simulations.
J Proteomics, 2020 Jan 16;211:103544. doi: 10.1016/j.jprot.2019.103544. Epub 2019 Nov 1.
PMID: 31683063
Blank-Landeshammer B, Richard VR, Mitsa G, Marques M, LeBlanc A, Kollipara L, Feldmann I, Couetoux du Tertre M, Gambaro K, McNamara S, Spatz A, Zahedi RP, Sickmann A, Batist G, Borchers CH.
Proteogenomics of Colorectal Cancer Liver Metastases: Complementing Precision Oncology with Phenotypic Data.
Cancer, 2019 Dec 1;11(12):1907. doi: 10.3390/cancers11121907.
PMID: 31805664
Popov KI, Makepeace KAT, Petrotchenko EV, Dokholyan NV, Borchers CH.
Insight into the Structure of the « Unstructured » Tau Protein.
Structure, 2019 Nov 5;27(11):1710-1715.e4. doi: 10.1016/j.str.2019.09.003. Epub 2019 Oct 15.
PMID: 31628033
Martínez de Paz A, Khajavi L, Martin H, Claveria-Gimeno R, Tom Dieck S, Cheema MS, Sanchez-Mut JV, Moksa MM, Carles A, Brodie NI, Sheikh TI, Freeman ME, Petrotchenko EV, Borchers CH, Schuman EM, Zytnicki M, Velazquez-Campoy A, Abian O, Hirst M, Esteller M, Vincent JB, Malnou CE, Ausió J.
MeCP2-E1 isoform is a dynamically expressed, weakly DNA-bound protein with different protein and DNA interactions compared to MeCP2-E2.
Epigenetics Chromatin, 2019 Oct 10;12(1):63. doi: 10.1186/s13072-019-0298-1.
PMID: 31601272
Bhardwaj M, Gies A, Weigl K, Tikk K, Benner A, Schrotz-King P, Borchers CH, Brenner H.
Evaluation and Validation of Plasma Proteins Using Two Different Protein Detection Methods for Early Detection of Colorectal Cancer.
Cancers (Basel), 2019 Sep 25;11(10):1426. doi: 10.3390/cancers11101426.
PMID: 31557860
Pires ES, Hardoim CCP, Miranda KR, Secco DA, Lobo LA, de Carvalho DP, Han J, Borchers CH, Ferreira RBR, Salles JF, Domingues RMCP, Antunes LCM.
The Gut Microbiome and Metabolome of Two Riparian Communities in the Amazon.
Front Microbiol, 2019 Sep 4;10:2003. doi:10.3389/fmicb.2019.02003. eCollection 2019.
PMID: 31555238
Sirois I, Aguilar-Mahecha A, Lafleur J, Fowler E, Vu V, Scriver M, Buchanan M, Chabot C, Ramanathan A, Balachandran B, Legare S, Przybytkowski E, Lan C, Krzemien U, Cavallone L, Aleynikova O, Ferrario C, Guilbert MC, Benlimame N, Saad A, Alaoui-Jamali M, Saragovi HU, Josephy S, O’Flanagan C, Hursting SD, Richard VR, Zahedi RP, Borchers CH, Bareke E, Nabavi S, Tonellato P, Roy JA, Robidoux A, Marcus EA, Mihalcioiu C, Majewski J, Basik M.
A unique morphological phenotype in chemoresistant triple negative breast cancer reveals metabolic reprogramming and PLIN4 expression as a molecular vulnerability.
Mol Cancer Res, 2019 Dec;17(12):2492-2507. doi: 10.1158/1541-7786.MCR-19-0264. Epub 2019 Sep 19.
PMID: 31537618
Oyama Y, Bartman CM, Bonney S, Lee JS, Walker LA, Han J, Borchers CH, Buttrick PM, Aherne CM, Clendenen N, Colgan SP, Eckle T.
Intense Light-Mediated Circadian Cardioprotection via Transcriptional Reprogramming of the Endothelium.
Cell Rep, 2019 Aug 6;28(6):1471-1484.e11. doi: 10.1016/j.celrep.2019.07.020.
PMID: 31390562
Mao F, Liu T, Hou X, Zhao H, He W, Li C, Jing Z, Sui J, Wang F, Liu X, Han J, Borchers CH, Wang JS, Li W.
Increased sulfation of bile acids in mice and human subjects with sodium taurocholate cotransporting polypeptide deficiency.
J Biol Chem, 2019 Aug 2;294(31):11853-11862. doi: 10.1074/jbc.RA118.007179. Epub 2019 Jun 14.
PMID: 31201272
Masson GR, Burke JE, Ahn NG, Anand GS, Borchers C, Brier S, Bou-Assaf GM, Engen JR, Englander SW, Faber J, Garlish R, Griffin PR, Gross ML, Guttman M, Hamuro Y, Heck AJR, Houde D, Iacob RE, Jørgensen TJD, Kaltashov IA, Klinman JP, Konermann L, Man P, Mayne L, Pascal BD, Reichmann D, Skehel M, Snijder J, Strutzenberg TS, Underbakke ES, Wagner C, Wales TE, Walters BT, Weis DD, Wilson DJ, Wintrode PL, Zhang Z, Zheng J, Schriemer DC, Rand KD.
Recommendations for performing, interpreting, and reporting hydrogen deuterium exchange mass spectrometry (HDX-MS) experiments.
Nat Methods, 2019 Jul;16(7):595-602. doi: 10.1038/s41592-019-0459-y. Epub 2019 Jun 27.
PMID: 31249422
Anwar MA, Dai DL, Wilson-McManus J, Smith D, Francis GA, Borchers CH, McManus BM, Hill JS, Cohen Freue GV.
Multiplexed LC/ESI-MRM-MS-based Assay for Identification of Coronary Artery Disease Biomarkers in Human Plasma.
Proteomics Clin Appl, 2019 Jul;13(4):e1700111. doi: 10.1002/prca.201700111. Epub 2019 Jan 28.
PMID: 30632678
Iacobucci C, Piotrowski C, Aebersold R, Amaral BC, Andrews PC, Bernfur K, Borchers CH, Brodie NI, Bruce JE, Cao Y, Chaignepain S, Chavez JD, Claverol S, Cox J, Davis TN, Degliesposti G, Dong MQ, Edinger N, Emanuelsson C, Gay M, Götze M, Gomes-Neto F, Gozzo FC, Gutierrez CB, Haupt C, Heck AJR, Herzog F, Huang L, Hoopmann MR, Kalisman N, Klykov O, Kukačka Z, Liu F, MacCoss MJ, Mechtler K, Mesika R, Moritz RL, Nagaraj N, Nesati V, Neves-Fereira AGC, Ninnis R, Novák P, O’Reilly FJ, Pelzing M, Petrotchenko EV, Piersimoni L, Plasencia M, Pukala TL, Rand KD, Rappsilber J, Reichman D, Sailer C, Sarnowski CP, Scheltema RA, Schmidt C, Schriemer DC, Shi Y, Skehel JM, Slavin M, Sobott F, Solis-Mezarino V, Stephanowitz H, Stengel F, Stieger CE, Trabjerg E, Trnka MJ, Vilaseca M, Viner R, Xiang Y, Yılmaz Ş, Zelter A, Ziemianowicz DS, Leitner A, Sinz A.
The First Community-Wide, Comparative Cross-linking Mass Spectrometry Study.
Anal Chem, 2019 Jun 4;91(11):6953-6961. doi: 10.1021/acs.analchem.9b00658. Epub 2019 May 22.
PMID: 31045356
Brodie NI, Popov KI, Petrotchenko EV, Dokholyan NV, Borchers CH.
Conformational ensemble of native α-synuclein in solution as determined by short-distance crosslinking constraint-guided discrete molecular dynamics simulations.
PLoS Comput Biol, 2019 Mar 27;15(3):e1006859. doi: 10.1371/journal.pcbi.1006859. eCollection 2019 Mar.
PMID: 30917118
Kashirina DN, Percy AJ, Pastushkova LK, Borchers CH, Kireev KS, Ivanisenko VA, Kononikhin AS, Nikolaev EN, Larina IM.
The molecular mechanisms driving physiological changes after long duration space flights revealed by quantitative analysis of human blood proteins.
BMC Med Genomics, 2019 Mar 13;12(Suppl 2):45. doi: 10.1186/s12920-019-0490-y.
PMID: 30871558
Teixeira F, Tse E, Castro H, Makepeace KAT, Meinen BA, Borchers CH, Poole LB, Bardwell JC, Tomás AM, Southworth DR, Jakob U.
Chaperone activation and client binding of a 2-cysteine peroxiredoxin.
Nat Commun, 2019 Feb 8;10(1):659. doi: 10.1038/s41467-019-08565-8.
PMID: 30737390
Shultis D, Mitra P, Huang X, Johnson J, Khattak NA, Gray F, Piper C, Czajka J, Hansen L, Wan B, Chinnaswamy K, Liu L, Wang M, Pan J, Stuckey J, Cierpicki T, Borchers CH, Wang S, Lei M, Zhang Y.
Changing the Apoptosis Pathway through Evolutionary Protein Design.
J Mol Biol, 2019 Feb 15;431(4):825-841. doi: 10.1016/j.jmb.2018.12.016. Epub 2019 Jan 6.
PMID: 30625288
Wang R, Sheps JA, Liu L, Han J, Chen PSK, Lamontagne J, Wilson PD, Welch I, Borchers CH, Ling V.
Hydrophilic bile acids prevent liver damage caused by lack of biliary phospholipid in Mdr2-/-mice.
J Lipid Res, 2019 Jan;60(1):85-97. doi: 10.1194/jlr.M088070. Epub 2018 Nov 11.
PMID: 30416103
Boczonadi V, King MS, Smith AC, Olahova M, Bansagi B, Roos A, Eyassu F, Borchers C, Ramesh V, Lochmüller H, Polvikoski T, Whittaker RG, Pyle A, Griffin H, Taylor RW, Chinnery PF, Robinson AJ, Kunji ERS, Horvath R.
Correction: Mitochondrial oxodicarboxylate carrier deficiency is associated with mitochondrial DNA depletion and spinal muscular atrophy-like disease.
Genet Med, 2019 Sep;21(9):2163-2164. doi: 10.1038/s41436-019-0506-1.
PMID: 31028354
Popp R, Li H, Borchers CH.
Immuno-MALDI (iMALDI) mass spectrometry for the analysis of proteins in signaling pathways.
Expert Rev Proteomics, 2018 Sep;15(9):701-708. doi: 10.1080/14789450.2018.1516147. Epub 2018 Sep 17.
PMID: 30169113
Han J, Higgins R, Lim MD, Atkinson K, Yang J, Lin K, Borchers CH.
Isotope-labeling derivatization with 3-nitrophenylhydrazine for LC/multiple-reaction monitoring-mass-spectrometry-based quantitation of carnitines in dried blood spots.
Anal Chim Acta, 2018 Dec 11;1037:177-187. doi: 10.1016/j.aca.2018.01.045. Epub 2018 Feb 5.
PMID: 30292292
Michaud SA, Sinclair NJ, Pětrošová H, Palmer AL, Pistawka AJ, Zhang S, Hardie DB, Mohammed Y, Eshghi A, Richard VR, Sickmann A, Borchers CH.
Molecular phenotyping of laboratory mouse strains using 500 multiple reaction monitoring mass spectrometry plasma assays.
Common Biol, 2018 Jun 27;1:78. doi: 10.1038/s42003-018-0087-6. eCollection 2018.
PMID: 30271959
Zahedi RP, Parker CE, Borchers CH.
Immuno-MALDI-TOF-MS in the Clinic.
Clin Chem, 2018 Sep;64(9):1271-1272. doi: 10.1373/clinchem.2018.292136. Epub 2018 Jul 17.
PMID: 30018057
Penn AM, Bibok MB, Saly VK, Coutts SB, Lesperance ML, Balshaw RF, Votova K, Croteau NS, Trivedi A, Jackson AM, Hegedus J, Klourfeld E, Yu AYX, Zerna C, Modi J, Barber PA, Hoag G, Borchers CH; SpecTRA study group.
Validation of a Proteomic Biomarker Panel to Diagnose Minor-Stroke and Transient Ischaemic Attack: Phase 2 of SpecTRA, a Large-Scale Translational Study.
Biomarkers, 2018 Dec;23(8):793-803. doi: 10.1080/1354750X.2018.1499130. Epub 2018 Aug 23.
PMID: 30010432
Wölter M, Okai CA, Smith D, Ruß M, Rath W, Pecks U, Borchers CH, Glocker MO.
Maternal Apolipoprotein B100 Serum Levels are Diminished in Pregnancies with Intrauterine Growth Restriction and Differentiate from Controls.
Proteomics Clin Appl, 2018 Nov;12(6):e1800017. doi: 10.1002/prca.201800017. Epub 2018 Jul 23.
PMID: 29956482
Mahdavi S, Jenkins DJA, Borchers CH, El-Sohemy A.
Genetic Variation in 9p21 and the Plasma Proteome.
J Proteome Res, 2018 Aug 3;17(8):2649-2656. doi: 10.1021/acs.jproteome.8b00117. Epub 2018 Jul 19.
PMID: 29905076
Mnatsakanyan R, Shema G, Basik M, Batist G, Borchers CH, Sickmann A, Zahedi RP.
Detecting post-translational modification signatures as potential biomarkers in clinical mass spectrometry.
Expert Rev Proteomics, 2018 Jun;15(6):515-535. doi: 10.1080/14789450.2018.1483340.
PMID: 29893147
Bhowmick P, Mohammed Y, Borchers CH.
MRMAssayDB: an integrated resource for validated targeted proteomics assays.
Bioinformatics, 2018 Oct 15;34(20):3566-3571. doi: 10.1093/bioinformatics/bty385.
PMID: 29762640
Popp R, Basik M, Spatz A, Batist G, Zahedi RP, Borchers CH.
How iMALDI can improve clinical diagnostics.
Analyst, 2018 May 15;143(10):2197-2203. doi: 10.1039/c8an00094h.
PMID: 29713694
Parker CE, Borchers CH.
The Special Issue: Clinical Proteomics for Precision Medicine.
Proteomics Clin Appl, 2018 Mar;12(2). doi: 10.1002/prca.201600144.
PMID: 29521035
Luehr TC, Koide EM, Wang X, Han J, Borchers CH, Helbing CC.
Metabolomic insights into the effects of thyroid hormone on Rana [Lithobates] catesbeiana metamorphosis using whole-body Matrix Assisted Laser Desorption/Ionization-Mass Spectrometry Imaging (MALDI-MSI).
Gen Comp Endocrinol, 2018 Sep 1;265:237-245. doi: 10.1016/j.ygcen.2018.02.012. Epub 2018 Feb 19.
PMID: 29470956
Liu T, Wang RX, Han J, Hao CZ, Qiu YL, Yan YY, Li LT, Wang NL, Gong JY, Lu Y, Zhang MH, Xie XB, Yang JC, You YJ, Li JQ, Knisely AS, Borchers CH, Ling V, Wang JS.
Comprehensive bile acid profiling in hereditary intrahepatic cholestasis: Genetic and clinical correlations.
Liver Int, 2018 Sep;38(9):1676-1685. doi: 10.1111/liv.13714. Epub 2018 Mar 12.
PMID: 29412511
Penn AM, Bibok MB, Saly VK, Coutts SB, Lesperance ML, Balshaw RF, Votova K, Croteau NS, Trivedi A, Jackson AM, Hegedus J, Klourfeld E, Yu AYX, Zerna C, Borchers CH.
Verification of a Proteomic Biomarker Panel to Diagnose Minor Stroke and Transient Ischaemic Attack: Phase 1 of SpecTRA, a Large-Scale Translational Study.
Biomarkers, 2018 May-Jun;23(4):392-405. doi: 10.1080/1354750X.2018.1434681. Epub 2018 Feb 12.
PMID: 29385837
Penn AM, Saly V, Trivedi A, Lesperance ML, Votova K, Jackson AM, Croteau NS, Balshaw RF, Bibok MB, Smith DS, Lam KK, Morrison J, Lu L, Coutts SB, Borchers CH.
Differential Proteomics for Distinguishing Ischemic Stroke from Controls: a Pilot Study of the SpecTRA Project.
Transl Stroke Res, 2018 Dec;9(6):590-599. doi: 10.1007/s12975-018-0609-z. Epub 2018 Jan 24.
PMID: 29368175
Dilworth D, Gudavicius G, Xu X, Boyce AKJ, O’Sullivan C, Serpa JJ, Bilenky M, Petrochenko EV, Borchers CH, Hirst M, Swayne LA, Howard P, Nelson CJ.
The prolyl isomerase FKBP25 regulates microtubule polymerization impacting cell cycle progression and genomic stability.
Nucleic Acids Res, 2018 Mar 16;46(5):2459-2478. doi: 10.1093/nar/gky008.
PMID: 29361176
Perzanowska A, Fatalska A, Wojtas G, Lewandowicz A, Michalak A, Krasowski G, Borchers CH, Dadlez M, Domanski D.
An MRM-Based Cytokeratin Marker Assay as a Tool for Cancer Studies: Application to Lung-Cancer Pleural Effusions.
Proteomics Clin Appl, 2018 Mar;12(2). doi: 10.1002/prca.201700084. Epub 2018 Feb 7.
PMID: 29352525
Brodie NI, Huguet R, Zhang T, Viner R, Zabrouskov V, Pan J, Petrotchenko EV, Borchers CH.
Top-down hydrogen-deuterium exchange analysis of protein structures using ultraviolet photodissociation (UVPD).
Anal Chem, 2018 Mar 6;90(5):3079-3082. doi: 10.1021/acs.analchem.7b03655. Epub 2018 Feb 1.
PMID: 29336549
Jardim A, Hardie DB, Boitz J, Borchers CH.
Proteomic Profiling of Leishmania donovani Promastigote Subcellular Organelles.
J Proteome Res, 2018 Mar 2;17(3):1194-1215. doi: 10.1021/acs.jproteome.7b00817. Epub 2018 Feb 5.
PMID: 29332401
Han J, Higgins R, Lim MD, Lin K, Yang J, Borchers CH.
Short-Term Stabilities of 21 Amino Acids in Dried Blood Spot.
Clin Chem, 2018 Feb;64(2):400-402. doi: 10.1373/clinchem.2017.278457. Epub 2017 Nov 2.
PMID: 29097508
Bowden JA, Heckert A, Ulmer CZ, Jones CM, Koelmel JP, Abdullah L, Ahonen L, Alnouti Y, Armando A, Asara JM, Bamba T, Barr JR, Bergquist J, Borchers CH, Brandsma J, Breitkopf SB, Cajka T, Cazenave-Gassiot A, Checa A, Cinel MA, Colas RA, Cremers S, Dennis EA, Evans JE, Fauland A, Fiehn O, Gardner MS, Garrett TJ, Gotlinger KH, Han J, Huang Y, Neo AH, Hyotylainen T, Izumi Y, Jiang H, Jiang H, Jiang J, Kachman M, Kiyonami R, Klavins K, Klose C, Kofeler HC, Kolmert J, Koal T, Koster G, Kuklenyik Z, Kurland IJ, Leadley M, Lin K, Maddipati KR, McDougall D, Meikle PJ, Mellett NA, Monnin C, Moseley MA, Nandakumar R, Oresic M, Patterson RE, Peake D, Pierce JS, Post M, Postle AD, Pugh R, Qui Y, Quehenberger O, Ramrup P, Rees J, Rembiesa B, Reynaud D, Roth MR, Sales S, Schuhmann K, Schwartzman ML, Serhan CN, Shevchenko A, Somerville SE, John-Williams LS, Surma MA, Takeda H, Thakare R, Thompson JW, Torta F, Triebl A, Trotzmuller M, Ubhayasekera SJK, Vuckovic D, Weir JM, Welti R, Wenk MR, Wheelock CE, Yao L, Yuan M, Zhao XH, Zhou S.
Harmonizing Lipidomics: NIST Interlaboratory Comparison Exercise for Lipidomics using Standard Reference Material 1950 Metabolites in Frozen Human Plasma.
J Lipid Res, 2017 Dec;58(12):2275-2288. doi: 10.1194/jlr.M079012. Epub 2017 Oct 6.
PMID: 28986437
Mohammed Y, Pan J, Zhang S, Han J, Borchers CH.
ExSTA: External Standard Addition Method for Accurate High-throughput Quantitation in Targeted Proteomics Experiments.
Proteomics Clin Appl, 2018 Mar;12(2):1600180. doi: 10.1002/prca.201600180. Epub 2017 Oct 25.
PMID: 28895300
Urao N, Mirza RE, Corbiere TF, Hollander Z, Borchers CH, Koh TJ.
Thrombospondin-1 and disease progression in dysferlinopathy.
Hum Mol Genet, 2017 Dec 15;26(24):4951-4960. doi: 10.1093/hmg/ddx378.
PMID: 29206970
Dilworth D, Upadhyay SK, Bonnafous P, Edoo AB, Bourbigot S, Pesek-Jardim F, Gudavicius G, Serpa JJ, Petrotchenko EV, Borchers CH, Nelson CJ, Mackereth CD.
The basic tilted helix bundle domain of the prolyl isomerase FKBP25 is a novel double-stranded RNA binding module.
Nucleic Acids Res, 2017 Nov 16;45(20):11989-12004. doi: 10.1093/nar/gkx852.
PMID: 29036638
Popp R, Li H, LeBlanc A, Mohammed Y, Aguilar-Mahecha A, Chambers AG, Lan C, Poetz O, Basik M, Batist G, Borchers CH.
Immuno-Matrix-Assisted Laser Desorption/Ionization Assays for Quantifying AKT1 and AKT2 in Breast and Colorectal Cancer Cell Lines and Tumors.
Anal Chem, 2017 Oct 3;89(19):10592-10600. doi: 10.1021/acs.analchem.7b02934. Epub 2017 Sep 20.
PMID: 28853539
Pedde RD, Li H, Borchers CH, Akbari M.
Microfluidic-Mass Spectrometry Interfaces for Translational Proteomics.
Trends Biotechnol, 2017 Oct;35(10):954-970. doi: 10.1016/j.tibtech.2017.06.006. Epub 2017 Jul 26.
PMID: 28755975
Richard VR, Domanski D, Percy AJ, Borchers CH.
An online 2D-reversed-phase – Reversed-phase chromatographic method for sensitive and robust plasma protein quantitation.
J Proteomics, 2017 Sep 25;168:28-36. doi: 10.1016/j.jprot.2017.07.018. Epub 2017 Jul 28.
PMID: 28757464
Peixoto RJM, Alves ES, Wang M, Ferreira RBR, Granato A, Han J, Gill H, Jacobson K, Lobo LA, Domingues RMCP, Borchers CH, Davies JE, Finlay BB, Antunes LCM.
Repression of Salmonella host cell invasion by aromatic small molecules from the human fecal metabolome.
Appl Environ Microbiol, 2017 Sep 15;83(19):e01148-17. doi: 10.1128/AEM.01148-17. Print 2017 Oct 1.
PMID: 28754707
Li H, Popp R, Frohlich B, Chen MX, Borchers CH.
Peptide and Protein Quantification Using Automated Immuno-MALDI (iMALDI).
J Vis Exp, 2017 Aug 18;(126):55933. doi: 10.3791/55933.
PMID: 28872133
Guarna MM, Hoover SE, Huxter E, Higo H, Moon KM, Domanski D, Bixby MEF, Melathopoulos AP, Ibrahim A, Peirson M, Desai S, Micholson D, White R, Borchers CH, Currie RW, Pernal SF, Foster LJ.
Peptide biomarkers used for the selective breeding of a complex polygenic trait in honeybees.
Sci Rep, 2017 Aug 21;7(1):8381. doi: 10.1038/s41598-017-08464-2.
PMID: 28827652
Larina IM, Percy AJ, Yang J, Borchers CH, M Nosovsky A, I Grigoriev A, N Nikolaev E.
Protein expression changes caused by spaceflight as measured for 18 Russian cosmonauts.
Sci Rep, 2017 Aug 15;7(1):8142. doi: 10.1038/s41598-017-08432-w.
PMID: 28811532
Brodie NI, Popov KI, Petrotchenko EV, Dokholyan NV, Borchers CH.
Solving protein structures using short-distance cross-linking constraints as a guide for discrete molecular dynamics simulations.
Science Advances, 2017 Jul 7;3(7):e1700479. doi: 10.1126/sciadv.1700479. eCollection 2017 Jul.
PMID: 28695211
LeBlanc A, Michaud SA, Percy AJ, Hardie DB, Yang J, Sinclair NJ, Proudfoot JI, Pistawka A, Smith DS, Borchers CH.
Multiplexed MRM-based Protein Quantitation Using Two Different Stable Isotope Labeled Peptides for Calibration.
J Proteome Res, 2017 Jul 7;16(7):2527-2536. doi: 10.1021/acs.jproteome.7b00094. Epub 2017 Jun 2.
PMID: 28516774
Wang X, Han J, Hardie DB, Yang J, Pan J, Borchers CH.
Metabolomic profiling of prostate cancer by matrix assisted laser desorption/ionization-Fourier transform ion cyclotron resonance mass spectrometry imaging using Matrix Coating Assisted by an Electric Field (MCAEF).
Biochim Biophys Acta, 2017 Jul;1865(7):755-767. doi: 10.1016/j.bbapap.2016.12.012. Epub 2016 Dec 23.
PMID: 28017863
Mohammed Y, van Vlijmen BJ, Yang J, Percy AJ, Palmblad M, Borchers CH, Rosendaal FR.
Multiplexed targeted proteomic assay to assess coagulation factor concentrations and thrombosis-associated cancer.
Blood Advances, 2017 Jun 20;1(15):1080-1087. doi: 10.1182/bloodadvances.2017007955. eCollection 2017 Jun 27.
PMID: 29296750
Li H, Han J, Pan J, Liu T, Parker CE, Borchers CH.
Current Trends in Quantitative Proteomics — an update.
J Mass Spectrom, 2017 May;52(5):319-341. doi: 10.1002/jms.3932.
PMID: 28418607
Review
Qiu YL, Gong JY, Feng JY, Wang RX, Han J, Liu T, Lu Y, Li LT, Zhang MH, Sheps JA, Wang NL, Yan YY, Li JQ, Chen L, Borchers CH, Sipos B, Knisely AS, Ling V, Xing QH, Wang JS.
Defects in MYO5B are associated with a spectrum of previously undiagnosed low γ-glutamyltransferase cholestasis.
Hepatology, 2017 May;65(5):1655-1669. doi: 10.1002/hep.29020. Epub 2017 Mar 23.
PMID: 28027573
Reynolds LA, Redpath SA, Yurist-Doutsch S, Gill N, Brown EM, van der Heijden J, Brosschot TP, Han J, Marshall NC, Woodward SE, Valdez Y, Borchers CH, Perona-Wright G, Finlay BB.
Enteric helminths promote Salmonella co-infection by altering the intestinal metabolome.
J Infect Dis, 2017 Apr 15;215(8):1245-1254. doi: 10.1093/infdis/jix141.
PMID: 28368463
Streijger F, Skinnider M, Rogalski JC, Balshaw R, Shannon CP, Prudova A, Belanger LM, Ritchie L, Tsang A, Christie S, Parent S, Mac-Thiong JM, Bailey C, Urquhart J, Ailon T, Paquette SJ, Boyd MC, Street J, Fisher CG, Dvorak MF, Borchers CH, Foster LJ, Kwon BK.
A Targeted Proteomics Analysis of Cerebrospinal Fluid after Acute Human Spinal Cord Injury.
J Neurotrauma, 2017 Jun 15;34(12):2054-2068. doi:10.1089/neu.2016.4879. Epub 2017 Apr 7.
PMID: 28276985
Percy AJ, Michaud SA, Jardim A, Sinclair NJ, Zhang S, Mohammed Y, Palmer AL, Hardie DB, Yang J, LeBlanc AM, Borchers CH.
Multiplexed MRM-based assays for the quantitation of proteins in mouse plasma and heart tissue.
Proteomics, 2017 Apr;17(7). doi: 10.1002/pmic.201600097. Epub 2016 Nov 18.
PMID: 27688154
Mohammed Y, Bhowmick P, Smith DS, Domanski D, Jackson AM, Michaud SA, Malchow S, Percy AJ, Chambers AG, Palmer A, Zhang S, Sickmann A, Borchers CH.
PeptideTracker: A knowledgebase for collecting and storing information on protein concentrations in biological tissues.
Proteomics, 2017 Apr;17(7). doi: 10.1002/pmic.201600210. Epub 2016 Dec 14.
PMID: 27683069
Sobsey CA, Froehlich B, Batist G, Borchers CH.
Immuno-MALDI-MS for Accurate Quantitation of Targeted Peptides from Volume-Restricted Samples.
In: Methods in Molecular Biology, 2515: 203-225 (2022).
Percy AJ, Borchers CH.
Detailed Method for Performing the ExSTA Approach in Quantitative Bottom-up Plasma Proteomics.
In: Methods in Molecular Biology, 2228: 353-384 (2021).
Parker CE, Warren Hines MRE, Mocanu V, Greer SF, Borchers CH.
Mass Spectrometric Determination of Protein Ubiquitination.
In: Methods in Molecular Biology, 1934: 191-221 (2019).
Petrotchenko EV, Serpa JJ, Borchers CH
Cross-linking Applications in Structural Proteomics.
In: Proteomics for Biological Discovery, Wiley, 2019, 175-196, Veenstra TD and JR Yates (Eds.).
Eshghi A, Borchers CH.
Multiple Reaction Monitoring Using Double Isotopologue Peptide Standards 6 for Protein Quantification.
In: Methods in Molecular Biology, 1788: 193-214 (2018).
Percy AJ, Yang J, Chambers AG, Mohammed Y, Miliotis, Borchers CH.
Protocol for Standardizing High-to-Moderate Abundance Protein Biomarker Assessments Through an MRM-with-Standard-Peptides Quantitative Approach.
In: Modern Proteomics – Sample Preparation, Analysis and Practical Applications, Advances in Experimental Medicine and Biology, Springer,
2016, Vol. 919, 515-531
Percy AJ, Yang J, Chambers AG, Borchers CH.
Increased Depth and Breadth of Plasma Protein Quantitation via Two-Dimensional Liquid Chromatography/Multiple Reaction Monitoring-Mass Spectrometry with Labeled Peptide Standards.
In: Methods in Molecular Biology, 1410: 1-21 (2016).
Han J, Parker, CE, Borchers CH.
Chemical derivatization to enhance metabolomic analysis by LC/ESI–MS.
In: Advanced LC-MS Applications in Metabolomics, Future-Science eBook, Han J, Parker, CE, Borchers CH, Borchers CH (Eds), 2015.
Percy AJ, Chambers AG, Parker CE, Borchers CH.
MRM-based Protein Quantification with Labeled Standards for Biomarker Discovery, Verification, and Validation in Human Plasma.
In: Quantitative Proteomics, Royal Society of Chemistry, 2014, Vol. 1, 316-325.
Parker CE, Domanski D, Percy AJ, Chambers AG, Camenzind AG, Smith DS, Borchers CH.
Mass Spectrometry in High-Throughput Clinical Biomarker Assays: Multiple Reaction Monitoring.
In: Topics in Current Chemistry, Springer, 2014, Vol. 336, 117-138.
Petrotchenko EV, Makepeace KAT, Serpa JJ. Borchers CH.
Analysis of protein Structure by Crosslinking Combined with Mass Spectrometry.
In: Methods in Molecular Biology, Humana Press, 2014, Vol. 1156, 447-463.
Percy AJ, Chambers AG, Parker CE, Borchers CH.
A Targeted Mass Spectrometric Approach for Quantitating Candidate Cancer Biomarker Proteins in Undepleted and Non-Enriched Human Plasma.
In: The Research and Biology of Cancer, iConcept book, 2014, Vol. 1, 33-48.
Chen YT, Parker CE, Chen HW, Chen CL, Domanski D, Smith DS, Wu CC, Chung T, Liang KH, Chen MC, Chang YS, Borchers CH, Yu JS.
Discovery and Validation Case Studies, Recommendations: A Pipeline that Integrates the Discovery and Verification Studies of Urinary Protein Biomarkers Reveals Candidate Markers for Bladder Cancer.
In: Comprehensive Biomarker Discovery and Validation for Clinical Application, Royal Society of Chemistry, 2013, Vol. 33, Chapter 10, 271-314.
Shah B, Reid JD, Kuzyk MA, Parker CE, Borchers CH.
Developing an iMALDI Method.
In: Methods in Molecular Biology, 1023: 97-120 (2013).
Kuzyk MA, Parker CE, Domanki D, Borchers CH.
Development of MRM-based Assays in Absolute Quantitation of Plasma Proteins.
In: Methods in Molecular Biology, Humana Press, 2013, Vol. 1023, 53-82.
Percy AJ, Chambers AG, Parker CE, Borchers CH.
Absolute Quantitation of Proteins in Human Blood by Multiplexed Multiple Reactions Monitoring Mass Spectrometry.
In: Methods in Molecular Biology, Humana Press, 2013, Vol.1000, 167-189.
Percy AJ, Chambers AG, Parker CE, Borchers CH.
Absolute quantitation of proteins in human blood by multiplexed reaction monitoring mass spectrometry.
In: Methods in Molecular Biology, 1000: 167-89 (2013).
Domanski D, Smith DS, Miller CA, Yang Y, Jackson AM, Cohen Freue G, Hill JS, Parker CE, Borchers CH.
High-Flow Multiplexed MRM-Based Analysis of Proteins in Human Plasma Without Depletion or Enrichment.
In: Clinical Mass Spectrometry, Saunders, 2011, Vol. 31, N. 3, 371-384.
Shah B, Kozlowski RL, Han J, Borchers CH.
Emerging Mass Spectrometry-Based Technologies for Analyses of Chromatin Changes: Analysis of Histones and Histone Modifications.
In: Methods in Molecular Biology, vol. 773, Editor: Allison R. Kernmode (ed), Seed Dormancy: Methods and Protocols, 2011, Vol. 773, 259-303.
Ohlund LB, Hardie DB, Elliott MH, Smith DS, Reid JD, Cohen-Freue GV, Bergman AP, Sasaki M, Robertson L, Balshaw RF, Ng RT, Mui A, McManus, BM, Keown PA, McMaster WR, Parker CE, Borchers CH.
Standard Operating Procedures and Protocols for the Preparation and Analysis of Plasma Samples using the iTRAQ Methodology.
In: Sample Preparation in Biological Mass Spectrometry, Editor: Ivanov A, and Lazarev A, Springer, New York, 2011, 575-624.
Ouvry-Patat SA, Torres MP, Gelfand CA, Quek HH, Easterling M, Speir JP, Borchers CH.
Top-down proteomics on a high-field Fourier transform ion cyclotron resonance mass spectrometer.
In: Methods in Molecular Biology, 492: 215-31 (2009).
Petrotchenko EV, Borchers CH.
Cross-Linking as a Tool Examine Protein Complexes: Examples of Crosslinking Strategies and Computational Modeling.
In: Mass Spectrometry Analysis For Protein-Protein Interactions and Dynamics, Editor: Chance M, John Willey & Sons, Inc. 2008, Chapter 8, 157-168.
Parker CE, Warren MR, Mocanu V, Greer SF, Borchers CH.
Mass spectrometric determination of protein ubiquitination.
In: Methods Molecular Biology, 2008, Vol. 446, 109-30.
Parker CE, Smith D, Suckau D, Borchers CH.
Mass Spectrometry-based Tissue Imaging.
In: Advanced Imaging in Biology and Medicine, Editor: Sensen CH, Hallgrimsson W, Benedikt, Springer New York, 2008, 131-146.
Robinette D, Neamati N, Tomer KB, Borchers CH.
Photoaffinity Labeling Combined with Mass Spectrometric Approaches as a Tool for Structural Proteomics.
In: Expert Review in Proteomics, 3(4): 399-408 (2006).
Parker CE, Mocanu V, Warren MR, Greer SF, Borchers CH.
Mass Spectrometric Determination of Protein Ubiquitination.
In: Methods in Molecular Biology, Editor: Patterson WC, Humana Press, NJ, 2005, Vol. 103, 153-173.
Parker CE, Warren MR, Loiselle D, Dechiva NN, Scarlett CO, Borchers CH.
Identification of Components of Protein Complexes.
In: Methods in Molecular Biology, Editor: Patterson WC, Humana Press, NJ, 2005, Vol. 103, 117-151.
Borchers CH, Chen T, Neamati N.
Application of Proteomics in Basic Biological Sciences and Cancer.
In: Molecular Carcinogenesis and the Molecular Biology of Human Cancer, Editor: Warshawsky D, Landolph JR, CRC, 200, 263-288.
Borchers C.
Automation and integration strategies in high-throughput genomics; Editorial advisory Board Member Commentaries.
In: High-Throughput Genomics: Maximizing Efficiency and Productivity for Drug Discovery and Development, Editor SJ, Walker TD, Howard GA,
Hawkins KE, Vrana JA, D. Lockwood & M.A. Branca, Cambridge Healthtech Institute, 2001,118-119.
Przybylski M, Borchers CH, Jetschke M, Schuhmacher R, Fiedler W, Mak, Glocker MO.
Molecular Approaches for the Characterization of Protein Tertiary Structures by Selective Chemical Modification and Mass Spectrometric Peptide Mapping.
In: Peptides-Chemistry, Structure and Biology, Editor: Hodges RS, Smith JA, Escom Science Publ., Leiden, 1994, 254-256.
Mina Farshadi, notre étudiante en MSc dans le laboratoire du Dr Borchers, s’est vue décerner le prix FeMS Empowerment 2023.
Nous sommes très fiers d’annoncer que notre scientifique, Deema Qasrawi, fera une présentation à la prochaine conférence MSACL 2023, 13e conférence annuelle et exposition à Monterey, CA. Sa présentation est intitulée « A simplified proteomics LC-MRM-MS assay for determination of ApoE genotypes in plasma samples ». Ne manquez pas d’assister à sa présentation orale en avril.
Nous sommes ravis de vous annoncer que notre technicien de laboratoire, Dennis Lee, a été accepté pour présenter un poster lors de la prochaine édition de MSACL 2023, 13e conférence annuelle et expositions à Monterey, CA. Sa présentation s’intitule « LC-MS assay for antibiotics in intensive care unit clinical setting« . Ne manquez pas de vous rendre à sa présentation par affiches en avril.
Le Dr Christoph Borchers a fait une présentation lors de la session petit-déjeuner d’Agilent Technologies à HUPO à Cancun. Il a parlé des nouvelles approches MRM pour une quantification plus rapide, plus multiplexée et plus sensible des protéines dans les matrices biologiques, y compris le sang et les tissus.
Deux de nos étudiants diplômés, Connie Sobsey et Georgia Mitsa, ont fait une présentation à HUPO à Cancun lors de la session Clinical Proteomics/Precision Medicine. La présentation de Georgia est intitulée « Clinical cancer research on metastases from refractory colorectal cancer using a multi-omics approach to improve current precision oncology treatment ». Ils ont identifié plusieurs nouvelles vulnérabilités thérapeutiques et des biomarqueurs putatifs pour mieux informer la prise de décision clinique. La présentation de Connie s’intitule « Targeted proteomics to verify the association between translational control pathways and capivasertib response in cancer cell lines and patient tumours ».
Nous accueillons un nouveau collègue, Yassene Mohammed, au Centre de protéomique du cancer Segal.
Nouvelle annonce de financement!
Génome Québec a annoncé en octobre les noms des 11 équipes de recherche des quatre établissements du Québec qui ont reçu plus de 4 millions de dollars en financement majeur en génomique. L’une des équipes sélectionnées est dirigée par le Dr Christoph Borchers.
Nous sommes très heureux d’annoncer que notre scientifique, Deema Qasrawi, fera une présentation orale lors de la prochaine conférence MANA à Edmonton, AB. Sa présentation est intitulée « Multiplexed dilute-and-shoot LC-MRM-MS clinical assay for determination of the metanephrines and catecholamines in human urine« . Ne manquez pas d’assister à sa présentation orale en septembre.
Félicitations au Centre de protéomique du cancer Segal pour le renouvellement du financement de la FCI-MSI pour le Centre d’innovation en métabolomique!
Consultez l’article complet pour savoir comment les principales installations de recherche nationales du Canada ont reçu des investissements importants pour stimuler la recherche et l’innovation.
Nous sommes très heureux d’annoncer que notre scientifique, Deema Qasrawi, fera une présentation orale lors de la 70e conférence de l’ASMS sur la spectrométrie de masse et les sujets connexes, qui se tiendra à Minneapolis (MN). Sa présentation est intitulée « Multiplexed dilute-and-shoot LC-MRM-MS clinical assay for determination of the metanephrines and catecholamines in human urine« . Ne manquez pas d’assister à sa présentation lors de la conférence de juin.
Vincent Lacasse, notre étudiant en doctorat du laboratoire du Dr Borchers, a remporté le prix du poster Gairdner au symposium CSHRF+Gairdner !
Nous avons des nouvelles passionnantes à partager! Le Dr René Zahedi et le Dr Christoph Borchers ont tous deux été récemment annoncés comme les nouveaux corédacteurs de la revue Expert Review of Proteomics..
Be sure to check out the International Conference on Multi-omics Technologies for Precision Medicine happening November 22-23, 2021. You’ll also want to attend the 10th Symposium on Structural Proteomics happening November 24-25, 2021!
Molecular and Cellular Proteomics a récemment mis en avant notre article « Proteomic Portraits Reveal Evolutionarily Conserved and Divergent Responses to Spinal Cord Injury » sur sa page web!
La dernière décennie a été riche en événements pour la protéomique, qui a connu un certain nombre de progrès, allant de nouveaux flux de travail et applications à des améliorations de l’instrumentation et des innovations bioinformatiques. Pour avoir une idée des principaux développements, nous avons demandé à des chercheurs de premier plan dans le domaine de nous donner leur avis sur les réalisations les plus remarquables en protéomique au cours des années 2010.
Le projet Megagrant du Center for Computational and Data-Intensive Science and Engineering a reçu un financement du ministère russe des sciences et de l’enseignement supérieur: Christoph Hermann Borchers et son nouveau projet de laboratoire « Next-Generation Proteomics to Improve Personalized Medicine and Health Care« . (Laboratoire de spectroscopie de masse), (durée 2 ans, 33 mln RUB/an).
Le groupe de l’UVic a contribué à 954 des plus de 2350 essais MRM ciblés sur l’homme et la souris au portail des essais du National Cancer Institute.
La candidate au doctorat Georgia Mitsa, du laboratoire de protéomique du Dr Christoph Borchers, a reçu deux prix de l’Université McGill :
Nous accueillons deux nouveaux collègues, Vanessa Gaspar qui travaille sur un projet financé par le FRQS et Evgeniy Petrotchenko qui est un expert en protéomique structurelle. N’oubliez pas de consulter la première partie de la série de webinaires omiques d’Agilent le 25 janvier.
Nous sommes fiers de faire partie du Consortium de Montréal sur le cancer (MCC) qui a récemment été financé par la Fondation Terry Fox et d’autres cofinanceurs. « En travaillant ensemble, les chercheurs du MCC viseront également à mieux comprendre comment divers aspects du système immunitaire sont liés à la leucémie aiguë et pourquoi la thérapie fonctionne pour certains patients mais pas pour d’autres. Ils espèrent identifier de nouveaux biomarqueurs et de nouvelles cibles qui répondront aux traitements d’immunothérapie. »
Nous sommes très fiers d’annoncer que notre scientifique, Deema Qasrawi, fera une présentation lors de la 13e conférence et exposition annuelle MSACL 2023 à Monterey, en Californie. 13th Annual Conference & Exhibits à Monterey, CA. Sa présentation est intitulée « Un essai simplifié de protéomique LC-MRM-MS pour la détermination des génotypes ApoE dans les échantillons de plasma”. Ne manquez pas d’assister à sa présentation orale en avril.

Nous sommes ravis de vous annoncer que notre technicien de laboratoire, Dennis Lee, a été accepté pour une présentation d’affiche à la prochaine MSACL 2023 13th Annual Conference & Exhibits à Monterey, CA. Sa présentation est intitulée « Essai LC-MS pour les antibiotiques dans le cadre clinique d’une unité de soins intensifs ». Ne manquez pas d’assister à sa présentation en avril.
Nous sommes très fiers d’annoncer que l’une de nos étudiantes en doctorat, Sahar Ibrahim, fera une présentation en personne lors de la prochaine conférence de l’ASMS sur la spectrométrie de masse et les sujets connexes. Sa présentation intitulée » Quantification précise de PTEN par immuno-MRM: un outil pour résoudre la controverse sur les biomarqueurs du cancer du sein » est basée sur la protéomique quantitative d’une protéine liée au cancer. Ne manquez pas sa remarquable présentation le jeudi 4 novembreth 2021.
Nous sommes ravis de vous annoncer que notre directeur associé, Evgeniy Petrotchenko, présentera un exposé en personne sur la protéomique structurelle intitulé » Découverte d’un nouveau site de liaison de l’ivacaftor à la protéine CFTR par marquage par photo-affinité combiné à la spectrométrie de masse ». lors de la conférence ASMS de cette année. Ne manquez pas de la suivre virtuellement ou en personne le 2021 novembre.
3755, chemin de la Côte Ste-Catherine
Montréal, Québec H3T 1E2
Pour toute question concernant la spectrométrie de masse analytique – protéomique et métabolomique, veuillez contacter :